
Guía del algoritmo de análisis de
imagen uPath HER2 (4B5) para mama


Tabla de contenido
Introducción 1
Resumen y explicación del algoritmo 2
Uso previsto 3
Uso previsto del producto 3
Objetivo de la Guía de algoritmos 3
Importancia clínica 4
Principios de la prueba 5
Limitaciones 6
Características de red 7
Seguridad de los datos 8
Flujo de trabajo del algoritmo uPath HER2 (4B5) 9
Flujo de trabajo del anatomopatólogo 11
Características de la tinción 18
Evaluación del anticuerpo VENTANA anti-HER2/neu (4B5) 18
Evaluación de la tinción del algoritmo uPath HER2 (4B5) 18
Características de rendimiento 30
Comparación de métodos 30
Estudios de reproducibilidad de anatomopatólogos 31
Estudios de reproducibilidad de los escáneres 32
Resolución de problemas 33
Referencias 37


Introducción
El Roche uPath enterprise software (uPath enterprise software)
con el algoritmo de análisis de imágenes de mama uPath HER2
(4B5) (algoritmo uPath HER2 (4B5)) es un sistema de software
diseñado como asistencia en la evaluación semicuantitativa de
la expresión de proteínas del receptor del factor de crecimiento
epidérmico 2 (HER2) humano en secciones histológicas
con tinción inmunohistoquímica (IHC) de tejidos normales y
neoplásicos fijados con formol y embebidos en parafina (FFPE).
El uPath enterprise software es una solución integral de software
de patología digital que permite a los laboratorios de patología
adquirir, administrar, observar, analizar, compartir e informar
sobre imágenes digitales de muestras de patología. Usando
el uPath enterprise software, el anatomopatólogo puede ver
imágenes digitales con diferentes aumentos, añadir anotaciones,
realizar mediciones de secciones de tejido, realizar análisis de
imágenes y generar informes.
Nota: el algoritmo de análisis de imágenes uPath HER2 (4B5) es
una metodología complementaria asistida por ordenador que
contribuye a la adquisición y medición de imágenes procedentes
de portaobjetos de vidrio relativas a muestras de tejido en las
que se ha llevado a cabo una tinción inmunohistoquímica para
detectar la presencia de la proteína HER2. Para garantizar
la validez de las puntuaciones del análisis de imágenes, es
responsabilidad del anatomopatólogo verificar la concordancia
y utilizar los controles adecuados tal y como se especifica en la
hoja de datos del anticuerpo VENTANA anti-HER2/neu (4B5)
(que se puede consultar en dialog.roche.com).
1 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Resumen y explicación del algoritmo
En el caso de las aplicaciones de análisis de imágenes, el
anatomopatólogo puede emplear uPath enterprise software para
seleccionar y destacar una o varias regiones de interés (ROI);
cada ROI se puede observar con varios aumentos y analizarse
después mediante el algoritmo uPath HER2 (4B5). Se genera
un recuento del número total de células tumorales diana y se
estratifican según estén teñidas o no. Las células teñidas se
estratifican según la intensidad de la tinción y la integridad de la
tinción de la membrana. El algoritmo uPath HER2 (4B5) combina
el porcentaje de tinción (con tinción, sin tinción), la intensidad
de la tinción (débil, media, fuerte) y la integridad de la tinción (sin
tinción, parcial, completa) para generar una puntuación HER2
en una escala de 0 a 3+. El algoritmo uPath HER2 (4B5) puede
generar una puntuación de un ROI en concreto o una puntuación
acumulada de todas las ROI seleccionadas en ese portaobjetos.
El anatomopatólogo puede aceptar la puntuación que facilita
el algoritmo uPath HER2 (4B5) o bien puede asignar una
puntuación diferente. El algoritmo uPath HER2 (4B5) no lleva a
cabo interpretaciones independientes de los datos y, por lo tanto,
solo un anatomopatólogo con formación debe utilizarlo junto
con el examen histológico, la información clínica importante
y los controles adecuados. El algoritmo uPath HER2 (4B5) se
ha diseñado y concebido para asistir al anatomopatólogo en la
evaluación de la expresión de la proteína HER2 en muestras de
tejido de mama teñidas con el anticuerpo VENTANA anti-HER2/
neu (4B5).
2 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Uso previsto del producto
El algoritmo de análisis de imágenes de mama uPath HER2 (4B5)
está previsto para su uso como ayuda para el anatomopatólogo
en la detección y medición semicuantitativa de la proteína HER2
en tejido mamario neoplásico fijado con formol y embebido en
parafina. Cuando se usa con VENTANA anti-HER2/neu (4B5)
Rabbit Monoclonal Primary Antibody (anticuerpo VENTANA
anti-HER2/neu (4B5)), está indicado para su uso como ayuda en
la evaluación de pacientes con cáncer de mama para quienes se
está considerando el tratamiento con Herceptin
®
(trastuzumab),
KADCYLA
®
(ado-trastuzumab emtansina) o PERJETA
®
(pertuzumab).
Nota: el algoritmo de análisis de imágenes uPath HER2 (4B5) es
una metodología complementaria asistida por ordenador que
contribuye a la adquisición y medición de imágenes procedentes
de portaobjetos de vidrio relativas a muestras de tejido en las
que se ha llevado a cabo una tinción inmunohistoquímica para
detectar la presencia de la proteína HER2. Para garantizar
la validez de las puntuaciones del análisis de imágenes, es
responsabilidad del anatomopatólogo verificar la concordancia
y utilizar los controles adecuados tal y como se especifica en la
hoja de datos del anticuerpo VENTANA anti-HER2/neu (4B5).
Este algoritmo es para uso diagnóstico in vitro (IVD).
Uso previsto
Objetivo de la Guía de algoritmos
Esta Guía del algoritmo uPath HER2 (4B5) (guía del algoritmo)
está concebida para:
• Proporcionar información básica sobre el uso previsto del
algoritmo uPath HER2 (4B5), los principios de la prueba y sus
limitaciones.
• Definir los materiales necesarios y los requisitos informáticos,
de seguridad de datos y de red.
• Mostrar las instrucciones paso a paso para ejecutar el algoritmo
uPath HER2 (4B5).
• Suministrar imágenes fotográficas que ilustren cómo se debe
utilizar el algoritmo uPath HER2 (4B5).
• Proporcionar a los anatomopatólogos una herramienta para
facilitar el uso del algoritmo uPath HER2 (4B5) en secciones de
mama FFPE teñidas con el anticuerpo VENTANA anti-HER2/neu
(4B5).
• Suministrar imágenes de ejemplo de casos difíciles para facilitar
orientación sobre cómo usar el algoritmo uPath HER2 (4B5) en
su evaluación.
• Mostrar las características de rendimiento del algoritmo uPath
HER2 (4B5).
3 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Importancia clínica
El cáncer de mama es el carcinoma más frecuente en las
mujeres y la segunda causa principal de muerte relacionada
con el cáncer.
1,2
En Norteamérica, las posibilidades de una
mujer de padecer cáncer de mama es de una entre ocho.
1
La
detección temprana y los tratamientos adecuados repercuten
significativamente en el índice de supervivencia global.
3,4
Las
muestras pequeñas de tejido son aptas para llevar a cabo análisis
rutinarios de inmunohistoquímica (IHC), motivo por el que este
método, junto con los anticuerpos que detectan los antígenos
relevantes para la interpretación del carcinoma, se ha convertido
en una herramienta eficaz para el diagnóstico y el pronóstico de
la enfermedad por parte del anatomopatólogo. Actualmente, la
oncoproteína HER2 es uno de los marcadores más importantes
del cáncer de mama.
5,6,7,8
Los fármacos de tratamiento Herceptin
(trastuzumab), KADCYLA (ado-trastuzumab emtansina) y
PERJETA (pertuzumab), han demostrado ser efectivos en
algunos pacientes con carcinoma de mama, dado que detienen
el crecimiento del cáncer y, en ciertos casos, lo invierten.
5,6,7,8,9
Los fármacos se basan en anticuerpos monoclonales
humanizados que se unen a la proteína HER2 en células
cancerosas.
5,8,9,10
Los métodos de diagnóstico in vitro orientados a la evaluación
del estado de HER2 en pacientes con cáncer de mama son
cruciales para asistir al facultativo en el establecimiento de un
tratamiento orientado a HER2.
5,6,7,8
El anticuerpo VENTANA
anti-HER2/neu (4B5) está indicado para su uso en la detección
mediante IHC de la expresión de la proteína HER2 en el cáncer
de mama. El algoritmo uPath HER2 (4B5) actúa como accesorio
del anticuerpo VENTANA anti-HER2/neu (4B5) como ayuda
en la evaluación de pacientes con cáncer de mama para los
que se contempla un tratamiento con Herceptin (trastuzumab),
con KADCYLA (ado-trastuzumab emtansina) o con PERJETA
(pertuzumab).
4 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Principios de la prueba
Criterios de intensidad y patrón de tinción de la membrana
celular para la asignación de la puntuación del algoritmo
uPath HER2 (4B5):
Patrón de tinción Puntuación Evaluación
de la tinción
HER2
No se observa tinción de la
membrana
0 Negativo
Tinción ligera y parcial de
la membrana en cualquier
proporción de las células
cancerosas
1+ Negativo
Tinción completa entre débil
y moderada de la membrana,
>10% de las células cancerosas
2+ Equívoca*
Tinción completa intensa de la
membrana, >10% de las células
cancerosas
3+ Positivo
* Se recomienda una prueba equivalente mediante ISH
uPath enterprise software con el algoritmo uPath HER2 (4B5)
emplea técnicas de análisis de imagen para obtener una
puntuación HER2.
El algoritmo uPath HER2 (4B5) utiliza parámetros predefinidos
para puntuar imágenes del tejido teñido con el anticuerpo
VENTANA anti-HER2/neu (4B5).
Pasos implicados en el análisis de imágenes:
• Detección de células en toda la imagen.
• Clasificación de células como células tumorales u otros tipos de
células.
• Identificación de la membrana teñida y estratificación de la
membrana en categorías: tinción parcial o completa y tinción de
intensidad débil, media o fuerte.
• Cálculo de la puntuación HER2 mediante la combinación de
la clasificación de las células, la intensidad de la tinción y la
clasificación de la membrana.
Cómo identifica el algoritmo de análisis de imágenes
HER2 las células tumorales y cómo se calcula la
puntuación:
• El algoritmo uPath HER2 (4B5) identifica las células tumorales
a través del color, la intensidad, el tamaño y las características
morfológicas.
• Las células tumorales que se han identificado se clasifican
como teñidas mediante la detección de la membrana y los
umbrales predefinidos.
• El algoritmo uPath HER2 (4B5) calcula la puntuación de HER2
mediante la identificación de las células con tinción y su
clasificación por intensidad e integridad de la tinción.
5 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

El algoritmo uPath HER2 (4B5) se ha diseñado para su uso con
el anticuerpo VENTANA anti-HER2/neu (4B5). Los resultados
de la prueba son tan buenos como la calidad y precisión del
portaobjetos IHC que se muestra en la imagen, y la imagen
posterior que se analiza.
El anatomopatólogo debe validar la sesión de tinción con el
anticuerpo VENTANA anti-HER2/neu (4B5) mediante un examen
microscópico manual de los portaobjetos de control HER2 para
verificar que se hayan obtenido los resultados previstos antes
de acceder a las imágenes de los portaobjetos en el uPath
enterprise software para su análisis.
Se deben seguir las recomendaciones del fabricante en cuanto
al anticuerpo VENTANA anti-HER2/neu (4B5), incluido el uso
de todos los materiales de control de calidad positivo y negativo
para cada sesión de tinción. Si los portaobjetos de control no
son aceptables con el examen microscópico manual, los tejidos
deben volver a teñirse con resultados aceptables.
El anatomopatólogo debe seguir las recomendaciones oportunas
durante la interpretación del anticuerpo VENTANA anti-HER2/
neu (4B5).
El algoritmo uPath HER2 (4B5) se ha diseñado para que sea
un anatomopatólogo con formación el que lo use junto con un
examen histológico, información clínica importante y controles
adecuados. No está diseñado para ser una herramienta
independiente; es necesaria la intervención humana competente
durante todo el proceso de análisis.
El algoritmo uPath HER2 (4B5) puede generar puntuaciones
incorrectas si las imágenes capturadas contienen una tinción
anómala (tinción nuclear, pigmentación, etc.).
El algoritmo uPath HER2 (4B5) rechazará los núcleos tumorales
alargados independientemente de la forma general de la célula.
Por esta razón, los tumores que contienen grandes cantidades
de células con núcleos tumorales alargados pueden precisar su
evaluación manual.
El algoritmo uPath HER2 (4B5) se ha preparado, desarrollado
y validado en: carcinomas lobulillares, ductales, mucinosos,
medulares, papilares y micropapilares invasivos, así como en
algunas lesiones metastásicas.
El algoritmo uPath HER2 (4B5) no se ha probado, como tampoco
se ha validado su seguridad y eficacia, cuando se utiliza con un
ordenador personal (PC) desde casa.
El algoritmo uPath HER2 (4B5) identifica las membranas teñidas
con DAB de cualquier intensidad e integridad. Utiliza un umbral
de ruido del 0,5 % de todas las células tumorales para puntuar
un caso como 1+. No sigue las directrices de la hoja de método
de usar un umbral de una celda para una puntuación de 1+.
En algunos casos, esto puede dar como resultado muy pocas
células que tengan tinción de la membrana puntuada como 0 en
lugar de 1+. Este umbral de ruido se estableció para garantizar
que los casos negativos verdaderos se puntúen con un 0.
El algoritmo uPath HER2 (4B5) se ha diseñado con sensibilidad
al color marrón (tinción DAB) para garantizar que se detecte
incluso una tinción de membrana débil. Por esta razón, puede
puntuar de forma incorrecta una muestra de tejido que
contenga artefactos marrones, como pigmentación, tinción de
fondo en fibras musculares o cualquier otro artefacto que esté
manchado de marrón. Dentro del uPath enterprise software
existen herramientas que permitirán al patólogo excluir dichas
áreas. Estas se describen con más detalle en la sección de
características de la tinción.
El algoritmo uPath HER2 (4B5) se ha concebido con sensibilidad
a los casos con puntuación 2+. Por este motivo, el algoritmo
uPath HER2 (4B5) puede asignar puntuaciones incorrectas de
2+ a determinados casos con puntuación 1+. En concreto, es
posible que se asigne una puntuación incorrecta de 2+ a ciertos
casos con puntuación 1+ y un porcentaje ligeramente inferior
al 10% de células tumorales con tinción completa o en casos
que contengan una gran cantidad de tinción citoplasmática en
células tumorales. En este determinado tipo de casos, puede ser
necesario que el anatomopatólogo anule la puntuación asignada.
La tinción citoplasmática se describe con más detalle en la
sección de características de la tinción.
Limitaciones
6 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

7 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama
Características de red
Se recomienda contar con una conexión de red de 1Gbps entre uPath enterprise software y el sistema de administración de imágenes
(IMS).
Tabla 1. Especificaciones del servidor de análisis de imágenes Roche uPath para laboratorios pequeños.
Parámetro Detalles
Procesador CPU @ 3,6GHz
Cantidad de núcleos 4
RAM 6x8GB (48GB)
Hyper-threading Desactivadas
Disco duro SSD de 240GB
Sistema operativo Microsoft Windows Server 2016
Soporte de máquina virtual (VM) Sí
Información adicional sobre VM El rendimiento puede ser diferente al de los servidores físicos por la sobrecarga de VM.
Antivirus Symantec Endpoint Protection Versión 12
Alimentación 110V/220V, 800vatios (2)
Puertos 2 USB, adaptador HPE Eth 10/25Gb
Alimentación ininterrumpida Recomendado
Tabla 2. Especificaciones del servidor de análisis de imágenes Roche uPath para laboratorios de medianos a grandes.
Parámetro Detalles
Procesador CPU @ 3,6GHz (2 procesadores)
Cantidad de núcleos 8 por procesador/16 en total
RAM 12x8GB (96 GB)
Hyper-threading Desactivadas
Disco duro SSD de 240GB (2 unidades)
Sistema operativo Microsoft Windows Server 2016
Soporte de máquina virtual (VM) Sí
Información adicional sobre VM El rendimiento puede ser diferente al de los servidores físicos por la sobrecarga de VM.
Antivirus Symantec Endpoint Protection Versión 12
Alimentación 110V/220V, 800vatios (2)
Puertos 2 USB, adaptador HPE Eth 10/25Gb
Alimentación ininterrumpida Recomendado

La presencia de software malintencionado o un acceso no
autorizado al instrumento puede dar lugar a una pérdida de
datos o dejar el instrumento inutilizable.
Para evitar infecciones por un software malintencionado, así
como para impedir accesos no autorizados y usos indebidos
del instrumento, es fundamental seguir las recomendaciones
siguientes:
• No instale ni ejecute ningún otro software en el instrumento.
• Asegúrese de que el resto de ordenadores y servicios de
la red están suficientemente protegidos contra software
malintencionado y cualquier acceso no autorizado. Estos
comprenden, entre otros, el sistema de información de
laboratorio (LIS), la memoria compartida de archivos de
almacenamiento, la memoria compartida de copias de
seguridad y los servicios.
• Los clientes son responsables de la seguridad de su red de
área local, sobre todo a la hora de protegerla contra software
y ataques malintencionados. Esta protección puede incluir
medidas como un firewall, que sirve para separar el dispositivo
de redes no controladas. Por otro lado, la protección también
puede comprender medidas que garanticen que la red
conectada está libre de códigos malintencionados.
• Limite el acceso físico al instrumento y a toda la infraestructura
informática conectada (ordenadores, cables, equipos de red, etc.)
• Asegúrese de que los archivos de almacenamiento y de copia
de seguridad del instrumento estén protegidos contra cualquier
acceso no autorizado y contra desastres potenciales. Esta lista
incluye la ubicación de almacenamiento remoto, los sitios de
detección de desastres y la transferencia segura de archivos de
copia de seguridad.
• En la medida de lo posible, utilice un firewall para restringir el
tráfico de la red.
• Las memorias USB se pueden usar para diversos tipos de copias
de seguridad y restauraciones. Una incorrecta manipulación de
una memoria USB puede provocar la pérdida de datos o el mal
funcionamiento del instrumento.
• Utilice solo las memorias USB que su representante de servicio
de Roche haya probado e instalado.
• En cada momento solo se puede utilizar un dispositivo USB.
Antes de insertar una memoria USB, verifique que no haya otro
dispositivo USB insertado.
Seguridad de los datos
• Antes de extraer una memoria USB, pulse el botón Expulsar de
Windows.
• No se debe modificar la configuración predeterminada del
sistema operativo (SO) proporcionada con el servidor, ya
que esto tiene implicaciones en las configuraciones de SO
reforzadas.
• Para evitar que un virus infecte el uPath enterprise software,
use la memoria USB exclusivamente en el instrumento. No
almacene otros datos en esta memoria USB.
8 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Flujo de trabajo del algoritmo uPath HER2 (4B5)
Material suministrado
• Algoritmo uPath HER2 (4B5)
Materiales necesarios pero no suministrados
• uPath enterprise software
• Portaobjetos de tejido mamario teñidos con el anticuerpo VENTANA anti-HER2/neu (4B5) mediante ultraView Universal DAB
Detection Kit en el instrumento BenchMark ULTRA.
• Escáner de portaobjetos VENTANA DP 200
Advertencias y precauciones
1. Para uso diagnóstico in vitro (IVD).
2. Solo para uso profesional.
3. PRECAUCIÓN: En los Estados Unidos, las normas nacionales restringen la venta de este dispositivo a médicos autorizados o por
orden de estos. (Rx Only)
4. Para informar sobre indicios de incidentes graves con respecto a este dispositivo, póngase en contacto con el representante local del
servicio técnico de Roche o con la autoridad competente del Estado Miembro o del país en el que se encuentra el usuario.
Flujo de trabajo
1. El tejido mamario en un portaobjetos de vidrio se tiñe con el anticuerpo VENTANA anti-HER2/neu (4B5) en un instrumento
BenchMark ULTRA.
2. La adquisición de imágenes (escaneado completo del portaobjetos) se realiza con el escáner de portaobjetos VENTANA DP 200 con
un aumento de 20 veces en un plano z.
3. Una vez que se adquieren las imágenes digitales, estas imágenes se transfieren desde el ordenador asociado con el escáner de
portaobjetos VENTANA DP 200 al sistema de administración de imágenes (IMS) en un servidor centralizado.
4. Después de la transferencia al servidor, se creará un caso en el uPath enterprise software. La creación de casos se puede producir
automáticamente a través de la comunicación con el sistema de información de laboratorio (LIS) con la información de identificación
(es decir, el tipo de tejido y el anticuerpo primario) que se encuentra en la etiqueta del código de barras del portaobjetos de
vidrio o que se introduce manualmente en uPath enterprise software. Consulte la Guía del usuario de uPath enterprise software
(PN1018943ES).
5. Al instalar el algoritmo uPath HER2 (4B5) (debe instalarse en un servidor independiente de uPath enterprise software y del IMS) y
acceder a una imagen de 20x con la tinción y el tipo de tejido adecuados, uPath enterprise software desencadena automáticamente el
Análisis Completo del Portaobjetos (Whole Slide Analysis, WSA).
6. WSA analiza automáticamente toda la imagen escaneada.
7. Una vez que finaliza el WSA, se informa desde uPath enterprise software al anatomopatólogo mediante una barra azul en la que se
puede leer “analysis successful” («análisis finalizado correctamente»).
8. El anatomopatólogo puede elegir ROI concretas para puntuarlas mediante las herramientas ROI y de Exclusion (Exclusión) que se
encuentran disponibles en uPath enterprise software.
9. El anatomopatólogo revisa los resultados del análisis de la imagen y acepta la puntuación o asigna manualmente una diferente.
9 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Tinción
• En la preparación y tinción del tejido se deben seguir las recomendaciones que se suministran en la hoja de datos del anticuerpo
VENTANA anti-HER2/neu (4B5).
• Todos los controles adecuados deben revisarse y los portaobjetos deben volver a teñirse si la tinción no cumple las directrices
descritas en la hoja de datos del anticuerpo VENTANA anti-HER2/neu (4B5).
• El algoritmo uPath HER2 (4B5) requiere el uso del anticuerpo VENTANA anti-HER2/neu (4B5) y cualquier material o suministros
adicionales que aparezcan recogidos en la hoja de datos del anticuerpo VENTANA anti-HER2/neu (4B5) para teñir los tejidos antes del
análisis.
• El anticuerpo VENTANA anti-HER2/neu (4B5) detecta la proteína HER2 en el tejido mamario FFPE teñido con ultraView Universal DAB
Detection Kit en un instrumento BenchMark ULTRA.
Captura de imágenes
Es necesario el uso del escáner de portaobjetos VENTANA DP 200 para escanear los portaobjetos. Las imágenes deben escanearse
con un aumento de 20x. Se recomienda que el tejido no tenga pliegues ni tinta. Si hay grandes secciones de la imagen desenfocadas,
se recomienda volver a escanear los portaobjetos. Para obtener más información sobre el escaneado, consulte la Guía de usuario del
escáner de portaobjetos para IVD VENTANA DP 200 (PN1017149ES).
Navegación general: uPath enterprise software
El uPath enterprise software está diseñado para adaptarse a las necesidades individuales y del sitio, incluidos, entre otros
configuración de informes e interfaz de usuario. Esta guía de algoritmos se centra en las herramientas necesarias para usar solamente
el algoritmo uPath HER2 (4B5). Para obtener más información sobre el uPath enterprise software, consulte la Guía del usuario de
uPath enterprise software.
10 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Flujo de trabajo del anatomopatólogo
Apertura de un caso en uPath enterprise software
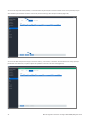
Para acceder a las imágenes del tejido mamario con tinción del anticuerpo VENTANA anti-HER2/neu (4B5), haga clic dos veces en un
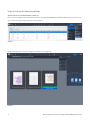
caso o selecciónelo y pulse la pestaña Viewer (Visor) (Figura1).
Aparecerá una pantalla con todas las imágenes asociadas a un caso (Figura2).
Figura 2
Figura 1
11 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

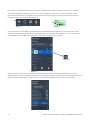
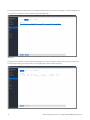
Una vez que se haya escaneado el portaobjetos de vidrio teñido con el anticuerpo VENTANA anti-HER2/neu (4B5) en un escáner
de portaobjetos VENTANA DP 200 a 20x, la imagen se importa a uPath enterprise software y se asocia con un caso. El algoritmo
uPath HER2 (4B5) activará automáticamente el WSA. El tiempo necesario para finalizar el paso de precálculo del WSA depende de
las especificaciones del servidor, los tamaños de imagen y el número de imágenes en la cola. Cuando se muestra, “waiting to start
auto-analysis” («esperando para iniciar el autoanálisis») quiere decir que las imágenes están en la cola y aún no se han analizado, y
aparecerá “analyzing” («analizando») cuando se realiza el WSA (Figuras3 y 4).
Una vez que la imagen se analiza por completo a través del WSA en uPath enterprise software, se mostrará el mensaje “analysis
successful” («análisis finalizado correctamente») debajo de la imagen del portaobjetos dentro del Viewer (Visor) (Figura5). Las
imágenes no se pueden puntuar antes de finalizar con éxito el WSA.
Figura 5
Figura 4
Figura 3
12 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

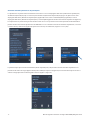
Dibujar el tumor completo (ROI): selección del área tumoral
Use el botón de la herramienta Freehand (Mano alzada) dentro del menú desplegable ROI (Figura 6) para seleccionar las áreas
tumorales en la imagen del portaobjetos IHC a analizar. La Figura 7 muestra una imagen que tiene una sola ROI dibujada. Se pueden
extraer ROI adicionales. Cada área seleccionada dará como resultado un ROI que aparecerá en el Slide Panel (Panel de portaobjetos)
(Figura 8).
Figura 7
Figura 8
Figura 6
13 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Dibujar la o las ROI del tumor completo: área de exclusión
Al dibujar la ROI, puede ser necesario excluir ciertas áreas; las áreas específicas que se deben evitar u omitir, así como los ejemplos,
se tratarán a continuación, en la sección de características de tinción. Use la herramienta de exclusión Freehand (Mano alzada)
dentro del menú desplegable Exclusion (Exclusión) (Figura 9) para excluir las áreas específicas (Figura 10). Si hay grandes áreas de la
imagen borrosas o desenfocadas, vuelva a escanear el portaobjetos.
Las áreas excluidas no se analizarán mediante el algoritmo uPath HER2 (4B5) y las células tumorales teñidas y sin tinción dentro de
esta área se excluirán del área de análisis total. Si ya se ha analizado la ROI y se realiza una exclusión, será necesario volver a analizar
la ROI y la superposición y la puntuación se actualizarán correctamente.
Dibujar una gran cantidad de exclusiones, especialmente las exclusiones Freehand (Mano alzada) complicadas, puede llevar mucho
tiempo y afectar a la eficacia del flujo de trabajo, aportando un impacto marginal a la puntuación final. Si un caso requiere una gran
cantidad de exclusiones, el anatomopatólogo debe hacer lo siguiente
• Dibujar múltiples ROI y excluir porciones de tejido que considere irrecuperables con un uso mínimo de la herramienta de Exclusion
(Exclusión).
• Limitar las exclusiones y anular manualmente la puntuación con otra puntuación.
Dibujar la o las ROI del tumor completo: supresión
Si una ROI del tumor completo seleccionada no es óptima, se puede eliminar. Seleccione la ROI del tumor completo haciendo clic en
el centro de la ROI en la imagen y después haciendo clic en el botón Delete (Eliminar) dentro del Slide Panel (Panel de portaobjetos)
(Figura 11) o dentro de la imagen del portaobjetos junto a la ROI (Figura 12). Aparecerá una ventana de confirmación. Seleccione
Confirm (Confirmar) para eliminar la ROI seleccionada. Seleccione Cancel (Cancelar) para conservar la ROI.
Figura 9
Figura 11
Figura 12
Figura 10
14 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Una vez que se han dibujado todas las áreas de exclusión y/o las ROI del tumor completo, la imagen está lista para ser analizada.
Seleccione la ROI del tumor completo haciendo clic en el centro de la ROI que se analizará o haciendo clic en la ROI en el Slide
Panel (Panel de portaobjetos). En cada ROI, seleccione el botón Image Analysis (Análisis de la imagen) del Slide Panel (Panel de
portaobjetos) (Figura13) o al lado de la ROI (Figura14).
Una vez que finaliza el análisis HER2, el resultado aparecerá en el Slide Panel (Panel de portaobjetos) en dos ubicaciones: debajo
de Slide Score (Puntuación de portaobjetos) y al lado de la ROI (Figura15). La puntuación de portaobjetos se basa en una suma del
estado de HER 2 en todos los ROI seleccionados, que es la puntuación que aparecerá en el informe.
También puede ver información más detallada en el menú desplegable Slide Score (Puntuación de portaobjetos) y en el menú
desplegable Details (Detalles) de la ROI haciendo clic en el icono desplegable (Figura16). Aparecerá el menú desplegable Slide Score
(Puntuación de portaobjetos) (Figura 17). Al hacer clic en el mismo icono desplegable también se oculta esta información.
Figura 15
Figura 17
Figura 16
Figura 13
Figura 14
15 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Análisis de imagen HER2: superposición en colores
Una vez que se ha pulsado el botón ROI(s) Analysis (Análisis de ROI) y se ha analizado el tejido, se mostrará una superposición de
colores en la ROI. En la imagen que aparece a continuación (Figura 18), la superposición en color rojo representa las membranas
celulares que se determinaron como teñidas positivamente para HER2. Al sujetar la imagen (pulsando el botón izquierdo del ratón y
moviendo la imagen) la superposición desaparece (Figura19). Cuando se deja de pulsar el botón del ratón, la superposición vuelve a
aparecer (Figura 18).
Figura 18
Figura 19
16 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Anulación manual de puntuaciones de portaobjetos
Las puntuaciones se pueden anular manualmente haciendo clic en el icono desplegable Slide Score (Puntuación de portaobjetos)
del Slide Panel (Portaobjetos) que se encuentra junto a Slide Score (Puntuación de portaobjetos) (Figura16). Aparecerá el menú
desplegable Slide Score (Puntuación de portaobjetos) (Figura 20). Al seleccionar el botón Edit (Editar) (Figura20) en el menú
desplegable Slide Score (Puntuación de portaobjetos), el anatomopatólogo puede introducir manualmente la puntuación (Figura21).
El campo Comments (Comentarios) permite introducir notas sobre el caso y/o la decisión de anular la puntuación automatizada. Se
pueden introducir manualmente puntuaciones de HER2 de 0 a 3+. Tras introducir una anulación manual de la puntuación, seleccione
el botón Confirm (Confirmación) (Figura 22). Aparecerá un mensaje de confirmación; haga clic en “Yes” («Sí»).
La puntuación que aparece dentro del Slide Panel (Panel de portaobjetos) reflejará ahora anulación manual de la puntuación. La
puntuación del análisis de imagen proporcionado junto a los ROI ya no aparecerá (Figura 23). El usuario tendrá la opción de volver a
analizar la imagen pulsando el botón del gráfico de barras (Figura 13, Figura 14).
Figura 23
Figura 21 Figura 22Figura 20
17 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Características de la tinción
Consulte la hoja de datos del anticuerpo VENTANA anti-HER2/neu (4B5) y la Guía de interpretación.
Evaluación del anticuerpo VENTANA anti-HER2/neu (4B5)
Se evalúan las células neoplásicas mamarias con tinción del anticuerpo VENTANA anti-HER2/neu (4B5) para determinar el
porcentaje, la integridad y la intensidad totales de la tinción de membrana de la célula tumoral con el fin de generar una puntuación
HER2, tal y como se describe en la Tabla de puntuación de HER2 en la sección anterior Principios de la prueba. La tinción
inmunohistoquímica en el cáncer de mama es membranosa y puede expresarse de forma homogénea o heterogénea a lo largo de la
neoplasia. La tinción citoplasmática de células tumorales no se incluye en la puntuación de HER2. La tinción de la membrana puede
tener patrón parcial o circunferencial. Se utiliza un anticuerpo de control negativo compatible con isotipo para evaluar la presencia de
fondo en las muestras de la prueba.
Evaluación de la tinción del algoritmo uPath HER2 (4B5)
El anatomopatólogo que vaya a utilizar el algoritmo uPath HER2 (4B5) debería estar familiarizado con la puntuación manual del
anticuerpo VENTANA anti-HER2/neu (4B5). El patólogo debe usar la herramienta Freehand (Mano alzada) para rodear toda el área
tumoral. El anatomopatólogo debe hacer referencia a los portaobjetos de control negativo y de la hematoxilina y la eosina asociados
antes de utilizar el algoritmo uPath HER2 (4B5). Al seleccionar las áreas a analizar, tenga en cuenta las limitaciones descritas en las
secciones Limitaciones y Áreas a evitar que aparecen a continuación. Si el anatomopatólogo no está de acuerdo con la puntuación
obtenida mediante el algoritmo uPath HER2 (4B5), debe anular manualmente la puntuación.
Entre los casos no evaluables se incluyen casos con tumor viable insuficiente, morfología inaceptable y antecedentes interferentes.
Los casos de cáncer de mama con suficientes células tumorales viables (en función de la puntuación del patólogo) y sin interferencias
del fondo en el portaobjetos HER2 IHC sí se aceptan para su evaluación.
18 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

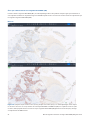
En las siguientes figuras (24-28) se proporcionan imágenes de diversos patrones de tinción y expresión. El nivel de expresión de HER2
se indica como un número entero (de 0 a 3+).
Figura24: imágenes escaneadas de tejido mamario teñido mediante IHC con una puntuación HER2 de 1+ en uPath
enterprise software; antes del análisis (arriba) y después del análisis (abajo). La superposición en color rojo representa la
membrana celular determinada para ser teñida positivamente para HER2.
19 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Figura25: imágenes escaneadas de tejido mamario teñido mediante IHC con una puntuación HER2 de 2+ en uPath
enterprise software; antes del análisis (arriba) y después del análisis (abajo). La superposición en color rojo representa la
membrana celular determinada para ser teñida positivamente para HER2.
20 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Figura26: imágenes escaneadas de tejido mamario teñido mediante IHC con una puntuación HER2 de 3+ en uPath
enterprise software; antes del análisis (arriba) y después del análisis (abajo). La superposición en color rojo representa la
membrana celular determinada para ser teñida positivamente para HER2.
21 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Figura27: imágenes escaneadas de un caso limítrofe con puntuación entre 1+ y 2+ en uPath enterprise software; antes del
análisis (arriba) y después del análisis (abajo). La superposición en color rojo representa la membrana celular determinada
para ser teñida positivamente para HER2. El algoritmo uPath HER2 (4B5) se ha concebido con sensibilidad a los casos con
puntuación 2+ y es posible que asigne puntuaciones incorrectas de 2+ a determinados casos de 1+. El anatomopatólogo
debe anular la puntuación en caso de ser necesario.
Casos limítrofes
22 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

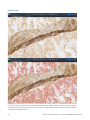
Figura28: ejemplo de tinción citoplasmática antes (arriba) y después del análisis (abajo). Es posible que se asigne una
puntuación incorrecta de 2+ a los casos que contengan una gran cantidad de tinción citoplasmática en las células tumorales.
En este tipo de casos, puede ser necesario que el anatomopatólogo anule la puntuación asignada.
Tinción citoplasmática
23 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Áreas que se deben evitar al usar el algoritmo uPath HER2 (4B5)
Cuando se utiliza el algoritmo uPath HER2 (4B5), los anatomopatólogos deben evitar puntuar cualquier región que normalmente se
evita al puntuar manualmente. La puntuación general de HER2 se puede anular si el usuario no está de acuerdo con la puntuación que
ha asignado el algoritmo uPath HER2 (4B5).
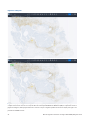
Necrosis
Figura29: ejemplo de tejido necrótico antes (arriba) y después del análisis (abajo). Los anatomopatólogos deben cumplir
las directrices descritas en la hoja de datos del anticuerpo VENTANA anti-HER2/neu (4B5). Es importante excluir el tejido
necrótico, dado que puede afectar a la forma en la que el algoritmo uPath HER2 (4B5) puntúa la muestra de tejido y dar lugar
a una puntuación de HER2 inexacta.
24 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Pliegues de tejido
Figura30: ejemplo de pliegues de tejido antes (arriba) y después del análisis (abajo). Los anatomopatólogos deben cumplir
las directrices descritas en la hoja de datos del anticuerpo VENTANA anti-HER2/neu (4B5). Es importante excluir los pliegues
de tejido, dado que pueden afectar a la forma en la que el algoritmo uPath HER2 (4B5) puntúa la muestra de tejido y dar lugar
a una puntuación de HER2 inexacta.
25 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Tinción de fondo
Figura 31: ejemplo de tinción de fondo antes (arriba) y después del análisis (abajo). Los anatomopatólogos deben cumplir las
directrices descritas en la hoja de datos del anticuerpo VENTANA anti-HER2/neu (4B5). Es importante excluir la tinción de
fondo, dado que puede afectar a la forma en la que el algoritmo uPath HER2 (4B5) puntúa la muestra de tejido y dar lugar a una
puntuación de HER2 inexacta.
26 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Figura32: ejemplo de tinta antes (arriba) y después del análisis (abajo). Los anatomopatólogos deben cumplir las directrices
descritas en la hoja de datos del anticuerpo VENTANA anti-HER2/neu (4B5). Es importante excluir la tinta, dado que puede
afectar a la forma en la que el algoritmo puntúa la muestra de tejido y dar lugar a una puntuación de HER2 inexacta.
Tinta
27 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Pigmentos endógenos
Figura33: ejemplo de pigmento endógeno antes (arriba) y después del análisis (abajo). Los anatomopatólogos deben
cumplir las directrices descritas en la hoja de datos del anticuerpo VENTANA anti-HER2/neu (4B5). Es importante excluir el
pigmento endógeno, dado que puede afectar a la forma en la que el algoritmo puntúa la muestra de tejido y dar lugar a una
puntuación de HER2 inexacta.
28 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Carcinoma ductal in situ (DCIS)
Figura34: ejemplo de DCIS antes (arriba) y después del análisis (abajo). Los anatomopatólogos deben cumplir las directrices
descritas en la hoja de datos del anticuerpo VENTANA anti-HER2/neu (4B5). Es importante excluir el DCIS, dado que puede
afectar a la forma en la que el algoritmo puntúa la muestra de tejido y dar lugar a una puntuación de HER2 inexacta.
29 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Características de rendimiento
Se evaluó el rendimiento del algoritmo uPath HER2 (4B5) mediante estudios de métodos de comparación, de concordancia entre
lectores, concordancia intralector, concordancia entre escáneres y concordancia intraescáner. Todos los análisis se llevaron a cabo
mediante el algoritmo uPath HER2 (4B5) en uPath enterprise software. Los anatomopatólogos que participaron siguieron las pautas
descritas en esta Guía de algoritmos en todos los estudios.
Comparación de métodos
Se llevó a cabo un estudio de comparación de métodos para comparar la puntuación obtenida mediante el algoritmo de análisis de
imágenes (IA) uPath HER2 (4B5) con las puntuaciones de los mismos casos que habían asignado los tres anatomopatólogos mediante
la lectura manual (MR). Se incluyeron un total de 529 evaluaciones de cada método en la comparativa. Los datos reales (GT),
obtenidos mediante el consenso del panel de anatomopatólogos, ninguno de los cuales había participado como anatomopatólogo
encargado de la lectura, sirvieron como referencia para calcular el porcentaje de concordancia positiva (PPA), el porcentaje de
concordancia negativa (NPA) y el porcentaje de concordancia global (OPA), tanto para IA como para MR. La diferencia en PPA, NPA y
OPA entre IA y MR también se facilita para mostrar la concordancia entre las puntuaciones de IA y MR. Los resultados del estudio, en
el que cada uno de los métodos se evaluó en comparación con los GT de referencia, demuestran que no el rendimiento del algoritmo
de IA uPath HER2 (4B5) no es inferior al de la MR de los anatomopatólogos.
Tabla 3. Concordancia: lectura manual frente a análisis de imagen
Estado de MR
Estado de los GT Estado del IA Concordancia Discordancia Total
Positivo
Concordancia 250 6 256
Discordancia 6 6 12
Total 256 12 268
PPA de MR n/N (%) (CI del 95%) 256/268 (95,5) (92,9, 97,8)
PPA de IA n/N (%) (CI del 95%) 256/268 (95,5) (92,9, 97,4)
Diferencia de PPA (IA-MR) n/N (%) (CI del 95%) 0/268 (0,0) (-3,0, 2,6)
Negativo
Concordancia 245 4 249
Discordancia 3 9 12
Total 248 13 261
NPA de MR n/N (%) (CI del 95%) 248/261 (95,0) (92,3, 97,5)
NPA de IA n/N (%) (CI del 95%) 249/261 (95,4) (93,1, 97,7)
Diferencia de NPA (IA-MR) n/N (%) (CI del 95%) 1/261 (0,4) (-1,1, 1,9)
Global
Concordancia 495 10 505
Discordancia 9 15 24
Total 504 25 529
OPA de MR n/N (%) (CI del 95%) 504/529 (95,3) (93,5, 97,2)
OPA de IA n/N (%) (CI del 95%) 505/529 (95,5) (93,6, 97,2)
Diferencia de OPA (IA-MR) n/N (%) (CI del 95%) 1/529 (0,2) (-1,5, 1,7)
Nota: Los intervalos de confianza del 95% bilaterales se calcularon mediante el método bootstrap percentil.
Nota: GT=Datos reales; IA=Análisis de imagen, MR=Lectura manual; CI=Intervalo de confianza
Nota: La concordancia y la discordancia hacen referencia al hecho de que los resultados de MR o de IA coincidan o no con los datos de GT.
Nota: En estos análisis se incluyen los resultados de los tres lectores anatomopatólogos.
30 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Estudios de reproducibilidad de anatomopatólogos
Los estudios de reproducibilidad de anatomopatólogos del algoritmo uPath HER2 (4B5) estaban formados por 529 evaluaciones
basadas en 180 casos independientes de cáncer de mama (cuya interpretación estuvo a cargo de tras anatomopatólogos con
formación) en el caso del estudio entre lectores y 118 evaluaciones basadas en 40 casos independientes de cáncer de mama (el mismo
anatomopatólogo fue el encargado de interpretar cada caso en tres ocasiones) en el caso del intralector.
Tabla 4. Concordancia interlectores del estado de HER2
Resultado modal Concordancia
Método Lector Resultado del
lector
Positivo Negativo Total Medida [a] % (n/N) CI del 95 % [b]
WTA IA
Global
Positivo 259 9 268 PPA 95,6 (259/271) (93,5, 97,4)***
Negativo 12 249 261 NPA 96,5 (249/258) (94,6, 98,4)***
Total 271 258 529 OPA 96,0 (508/529) (94,7, 97,4)***
Lector 1
Positivo 81 0 81 PPA 89,0 (81/91) (80,9, 93,9)**
Negativo
10 86 96 NPA
100,0 (86/86) (95,7, 100,0)**
Total 91 86 177 OPA 94,4 (167/177) (89,9, 96,9)**
Lector 2
Positivo
88 7 95 PPA
98,9 (88/89) (93,9, 99,8)**
Negativo 1 79 80 NPA 91,9 (79/86) (84,1, 96,0)**
Total 89 86 175 OPA 95,4 (167/175) (91,2, 97,7)**
Lector 3
Positivo 90 2 92 PPA 98,9 (90/91) (94,0, 99,8)**
Negativo 1 84 85 NPA 97,7 (84/86) (91,9, 99,4)**
Total 91 86 177 OPA 98,3 (174/177) (95,1, 99,4)**
WTA=Toda el área tumoral, AI=Análisis de imagen; CI=Intervalo de confianza
[a] PPA = Porcentaje de concordancia positiva; NPA = Porcentaje de concordancia negativa; OPA = Porcentaje de concordancia global.
[b] Los intervalos de confianza se calcularon utilizando el método de muestreo porcentual (***) o el método de Wilson (**).
Nota: en el análisis solo se incluyeron observaciones con evaluaciones presentes.
Tabla 5. Concordancia intralectores del estado de HER2
Resultado modal Concordancia
Método Lectura Resultado del
lector
Positivo Negativo Total Medida [a] % (n/N) CI del 95 % [b]
WTA IA
Global
Positivo 69 0 69 PPA 95,8 (69/72) (91,7, 100,0)***
Negativo 3 46 49 NPA 100,0 (46/46) (92,3, 100,0)**
Total 72 46 118 OPA 97,5 (115/118) (94,8, 100,0)***
Lectura 1
Positivo 21 0 21 PPA 87,5 (21/24) (69,0, 95,7)**
Negativo 3 16 19 NPA 100,0 (16/16) (80,6, 100,0)**
Total 24 16 40 OPA 92,5 (37/40) (80,1, 97,4)**
Lectura 2
Positivo
24 0 24 PPA
100,0 (24/24) (86,2, 100,0)**
Negativo 0 15 15 NPA 100,0 (15/15) (79,6, 100,0)**
Total 24 15 39 OPA 100,0 (39/39) (91,0, 100,0)**
Lectura 3
Positivo 24 0 24 PPA 100,0 (24/24) (86,2, 100,0)**
Negativo 0 15 15 NPA 100,0 (15/15) (79,6, 100,0)**
Total 24 15 39 OPA 100,0 (39/39) (91,0, 100,0)**
WTA=Toda el área tumoral, AI=Análisis de imagen; CI=Intervalo de confianza
[a] PPA = Porcentaje de concordancia positiva; NPA = Porcentaje de concordancia negativa; OPA = Porcentaje de concordancia global.
[b] Los intervalos de confianza se calcularon utilizando el método de muestreo porcentual (***) o el método de Wilson (**).
Nota: en el análisis solo se incluyeron observaciones con evaluaciones presentes.
31 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Estudios de reproducibilidad de los escáneres
Los estudios de reproducibilidad de los escáneres del algoritmo uPath HER2 (4B5) estuvo formado por 360 evaluaciones basadas
en 120 casos independientes de cáncer de mama. Un usuario formado los escaneó tres veces en cada uno de los tres escáneres de
portaobjetos VENTANA DP 200 a 20X y los analizó mediante el algoritmo uPath HER2 (4B5). Las puntuaciones se compararon entre
escáneres.
Tabla 6. Concordancia entre escáneres del estado de HER2
Resultado modal Concordancia
Método Escáner Resultado del
escáner
Positivo Negativo Total Medida [a] % (n/N) CI del 95 % [b]
WTA IA
Global
Positivo 230 0 230 PPA 98,3 (230/234) (95,7, 100,0)***
Negativo 4 126 130 NPA 100,0 (126/126) (97,0, 100,0)**
Total 234 126 360 OPA 98,9 (356/360) (97,2, 100,0)***
Escáner 1
Positivo 77 0 77 PPA 98,7 (77/78) (96,2, 100,0)***
Negativo
1 42 43 NPA
100,0 (42/42) (91,6, 100,0)**
Total 78 42 120 OPA 99,2 (119/120) (97,5, 100,0)***
Escáner 2
Positivo
76 0 76 PPA
97,4 (76/78) (92,3, 100,0)***
Negativo 2 42 44 NPA 100,0 (42/42) (91,6, 100,0)**
Total 78 42 120 OPA 98,3 (118/120) (95,0, 100,0)***
Escáner 3
Positivo 77 0 77 PPA 98,7 (77/78) (96,2, 100,0)***
Negativo 1 42 43 NPA 100,0 (42/42) (91,6, 100,0)**
Total 78 42 120 OPA 99,2 (119/120) (97,5, 100,0)***
WTA=Toda el área tumoral, AI=Análisis de imagen; CI=Intervalo de confianza
[a] PPA = Porcentaje de concordancia positiva; NPA = Porcentaje de concordancia negativa; OPA = Porcentaje de concordancia global.
[b] Los intervalos de confianza se calcularon utilizando el método de muestreo porcentual (***) o el método de Wilson (**). Dado que se combinaron los datos de
las tres sesiones en cada escáner, se recomienda llevar a cabo los cálculos del intervalo de confianza mediante el método bootstrap. Si se obtiene un resultado de
concordancia del 100%, se aconseja utilizar el método Wilson, dado que el método bootstrap no suministrará intervalos de confianza razonables.
Nota: en el análisis solo se incluyeron observaciones con evaluaciones presentes.
Nota: En el análisis se combinaron los datos de las tres sesiones de cada escáner.
Tabla 7. Concordancia intraescáner del estado de HER2
Resultado modal Concordancia
Método Escáner Resultado del
escáner
Positivo Negativo Total Medida [a] % (n/N) CI del 95 % [b]
WTA IA
Global
Positivo 229 1 230 PPA 99,1 (229/231) (97,9, 100,0)***
Negativo 2 128 130 NPA 99,2 (128/129) (97,4, 100,0)***
Total 231 129 360 OPA 99,2 (357/360) (97,8, 100,0)***
Escáner 1
Positivo 77 0 77 PPA 98,7 (77/78) (96,2, 100,0)***
Negativo
1 42 43 NPA
100,0 (42/42) (91,6, 100,0)**
Total 78 42 120 OPA 99,2 (119/120) (97,5, 100,0)***
Escáner 2
Positivo
75 1 76 PPA
100,0 (75/75) (95,1, 100,0)**
Negativo 0 44 44 NPA 97,8 (44/45) (93,3, 100,0)***
Total 75 45 120 OPA 99,2 (119/120) (97,5, 100,0)***
Escáner 3
Positivo 77 0 77 PPA 98,7 (77/78) (96,2, 100,0)***
Negativo 1 42 43 NPA 100,0 (42/42) (91,6, 100,0)**
Total 78 42 120 OPA 99,2 (119/120) (97,5, 100,0)***
WTA=Toda el área tumoral, AI=Análisis de imagen; CI=Intervalo de confianza
[a] PPA = Porcentaje de concordancia positiva; NPA = Porcentaje de concordancia negativa; OPA = Porcentaje de concordancia global.
[b] Los intervalos de confianza se calcularon utilizando el método de muestreo porcentual (***) o el método de Wilson (**). Dado que se combinaron los datos de
las tres sesiones en cada escáner, se recomienda llevar a cabo los cálculos del intervalo de confianza mediante el método bootstrap. Si se obtiene un resultado de
concordancia del 100%, se aconseja utilizar el método Wilson, dado que el método bootstrap no suministrará intervalos de confianza razonables.
Nota: en el análisis solo se incluyeron observaciones con evaluaciones presentes.
Nota: En el análisis se combinaron los datos de las tres sesiones de cada escáner.
32 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Resolución de problemas
Volver a generar un análisis fallido
Si se produce un error al realizar el WSA en el uPath enterprise software, se mostrará “analysis error” («error en el análisis») debajo de
la imagen del portaobjetos en una barra de color rojo (Figura 35).
Para volver a generar un análisis en uPath enterprise software, inicie sesión como Administrator (Administrador) (Figura36).
Una vez dentro de Administrator Settings (Ajustes de administrador), haga clic en Job Queue (Cola de trabajos) (Figura 37).
Figura 35
Figura 36
Figura 37
33 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Al seleccionar la pestaña Failed (Fallido), se mostrarán todos los portaobjetos con análisis fallidos. Seleccione el portaobjetos que
debe analizarse para habilitar los botones Cancel Job (Cancelar trabajo) y Start Job (Iniciar trabajo) (Figura 38).
Al seleccionar Start Job (Iniciar trabajo), se iniciará el análisis y, si tiene éxito, se mostrará “Job scheduled successfully” («Trabajo
planificado correctamente») en la parte superior de la pantalla en una barra de color verde (Figura 39).
Figura 38
Figura 39
34 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

La imagen del portaobjetos aparecerá ahora en la página Pending (Pendiente). Una vez en esta página, si hay varias imágenes en la
cola, es posible reorganizar los trabajos por orden de prioridad (Figura40).
Después de unos segundos, la imagen superior del portaobjetos se moverá a la página In-Progress (En progreso) y mostrará una
barra de progreso. No es necesario permanecer en esta página para realizar el análisis (Figura 41).
Figura 41
Figura 40
35 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

Cuando finaliza un análisis de portaobjetos, se moverá a la página Canceled/Complete (Cancelado/Completo) (Figura 42). En el
Viewer (Visor) de uPath, aparecerá el mensaje “analysis successful” («análisis finalizado correctamente») debajo de la imagen del
portaobjetos (Figura43) y todas las funciones analíticas importantes para el portaobjetos quedarán disponibles (Figura41).
Figura 42
Figura 43
36 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama

37 Guía del algoritmo de análisis de imagen uPath HER2 (4B5) para mama
Referencias
1. Hsu JL, Hung MC. The Role of HER2, EGFR, and Other Receptor Tyrosine Kinases
in Breast Cancer. Cancer Metastasis Rev. 2016;35(4):575-588.
2. Dickson RB, and Lippman ME. Genes, Oncogenes, and Hormones. Boston: Kluwer Academic Publishers; 1992.
3. Herceptin (Trastuzamab) [Package Insert]. EMEA (European Medicines Agency). http://www.ema.europa.eu/docs/
en_GB/document_library/EPAR_-_Product_Information/human/000278/WC500074922.pdf. Published 01/03/2010.
4. Roche PC. Immunohistochemical stains for breast cancer. Mayo Clin Proc. 1994;69: 57-58.
5. Slamon DJ, Leyland-Jones B, Shak S, et al. Use of Chemotherapy Plus a Monoclonal Antibody against HER2
for Metastatic Breast Cancer That Overexpresses HER2. N Engl J Med. 2001;344(11):783-792.
6. Swain SM, Kim SB, Cortés J, Ro J, Semiglazov V, Campone M, Ciruelos E, Ferrero JM, Schneeweiss A,
Knott A, Clark E, Ross G, Benyunes MC, Baselga J. Pertuzumab, trastuzumab, and docetaxel for HER2-
positive metastatic breast cancer (CLEOPATRA study): overall survival results from a randomised,
double-blind, placebo-controlled, phase 3 study. Lancet Oncol. 2013 May;14(6):461-71.
7. Hurvitz SA, Dirix L, Kocsis J, Bianchi GV, Lu J, Vinholes J, Guardino E, Song C, Tong B, Ng V, Chu YW, Perez EA.
Phase II randomized study of trastuzumab emtansine versus trastuzumab plus docetaxel in patients with human
epidermal growth factor receptor 2-positive metastatic breast cancer. J Clin Oncol. 2013 Mar 20;31(9):1157-63.
8. von Minckwitz G, Huang CS, Mano MS, et al. Trastuzumab Emtansine for Residual Invasive
HER2-Positive Breast Cancer. N Engl J Med. 2019;380(7):617-628.
9. Moasser MM, Krop IE. The Evolving Landscape of HER2 Targeting in Breast Cancer. JAMA Oncol. 2015;1(8):1154-1161.
10. Hudis CA. Trastuzumab--Mechanism of Action and Use in Clinical Practice. N Engl J Med. 2007;357(1):39-51.

Información de contacto
Roche Diagnostics GmbH
Sandhofer Strasse 116
D-68305 Mannheim
Germany
+800 5505 6606
dialog.roche.com
© 2020 Ventana Medical Systems, Inc. y Roche Diagnostics
International, Inc. Todos los derechos reservados.
VENTANA, BENCHMARK, ultraView, UPATH y el logotipo de
VENTANA son marcas comerciales de Roche. Todas las demás
marcas comerciales pertenecen a sus respectivos propietarios.
Todas las incorporaciones, eliminaciones y modificaciones están
indicados con una barra de cambios en el margen.
1019178ES Rev A
2020-12-18
09103104001
-
 1
1
-
 2
2
-
 3
3
-
 4
4
-
 5
5
-
 6
6
-
 7
7
-
 8
8
-
 9
9
-
 10
10
-
 11
11
-
 12
12
-
 13
13
-
 14
14
-
 15
15
-
 16
16
-
 17
17
-
 18
18
-
 19
19
-
 20
20
-
 21
21
-
 22
22
-
 23
23
-
 24
24
-
 25
25
-
 26
26
-
 27
27
-
 28
28
-
 29
29
-
 30
30
-
 31
31
-
 32
32
-
 33
33
-
 34
34
-
 35
35
-
 36
36
-
 37
37
-
 38
38
-
 39
39
-
 40
40
-
 41
41
-
 42
42
Artículos relacionados
-
Roche uPath PD-L1 image analysis for NSCLC IVD Algorithm Manual de usuario
-
Roche VENTANA anti-ALK D5F3 Primary Antibody Manual de usuario
-
Roche uPath IVD Manual de usuario
-
Roche iScan HT RUO Manual de usuario
-
Roche Virtuoso Manual de usuario
-
Roche Virtuoso Manual de usuario
-
Roche BenchMark XT/LT Interpretation Guide
-
Roche VENTANA PD-L1 (SP263) Assay, Universal, CE IVD, Predictive, NSCLC Interpretation Guide
Otros documentos
-
 Hologic Genius Review Station El manual del propietario
Hologic Genius Review Station El manual del propietario
-
 Monacor 25.7160 El manual del propietario
Monacor 25.7160 El manual del propietario
-
 Hologic ThinPrep 5000 Processor El manual del propietario
Hologic ThinPrep 5000 Processor El manual del propietario
-
 Hologic Genius Digital Diagnostics System Instrucciones de operación
Hologic Genius Digital Diagnostics System Instrucciones de operación
-
3B SCIENTIFIC L51 Manual de usuario
-
 Hologic ThinPrep 2000 Processor Instrucciones de operación
Hologic ThinPrep 2000 Processor Instrucciones de operación
-
 Hologic Quantra Guía del usuario
Hologic Quantra Guía del usuario
-
 Hologic ThinPrep Genesis Processor Instrucciones de operación
Hologic ThinPrep Genesis Processor Instrucciones de operación
















































